
原文标题:REASONING-ENHANCED HEALTHCARE PREDICTIONS WITH KNOWLEDGE GRAPH COMMUNITY RETRIEVAL
原文作者:Pengcheng Jiang, Cao Xiao, Minhao Jiang, Parminder Bhatia, Taha Kass-Hout, Jimeng Sun, Jiawei Han原文链接:https://openreview.net/forum?id=8fLgt7PQza发表会议:ICLR"25笔记作者:彭佳仁@安全学术圈主编:黄诚@安全学术圈编辑:张贝宁@安全学术圈
1. 研究背景
传统的检索增强生成(RAG)方法常常检索到稀疏或不相关的信息,从而影响了大语言模型(LLMs)进行预测的准确性。
论文引入了 KARE,一个新颖的框架,它将知识图谱(KG)的社区级检索与 LLM 的推理相结合,以增强医疗保健预测。 KARE 通过整合生物医学数据库、临床文献和 LLM 生成的见解,构建了一个全面的多源知识图谱,并利用层次化图社区检测和摘要技术对其进行组织,以实现精确且与上下文相关的知识检索。
论文的主要创新包括:(1) 一种密集的医学知识结构化方法,能够准确检索相关信息;(2) 一种动态知识检索机制,通过集中的、多方面的医学见解来丰富患者的上下文信息;以及 (3) 一个推理增强的预测框架,该框架利用这些丰富的上下文信息,生成准确且可解释的临床预测。
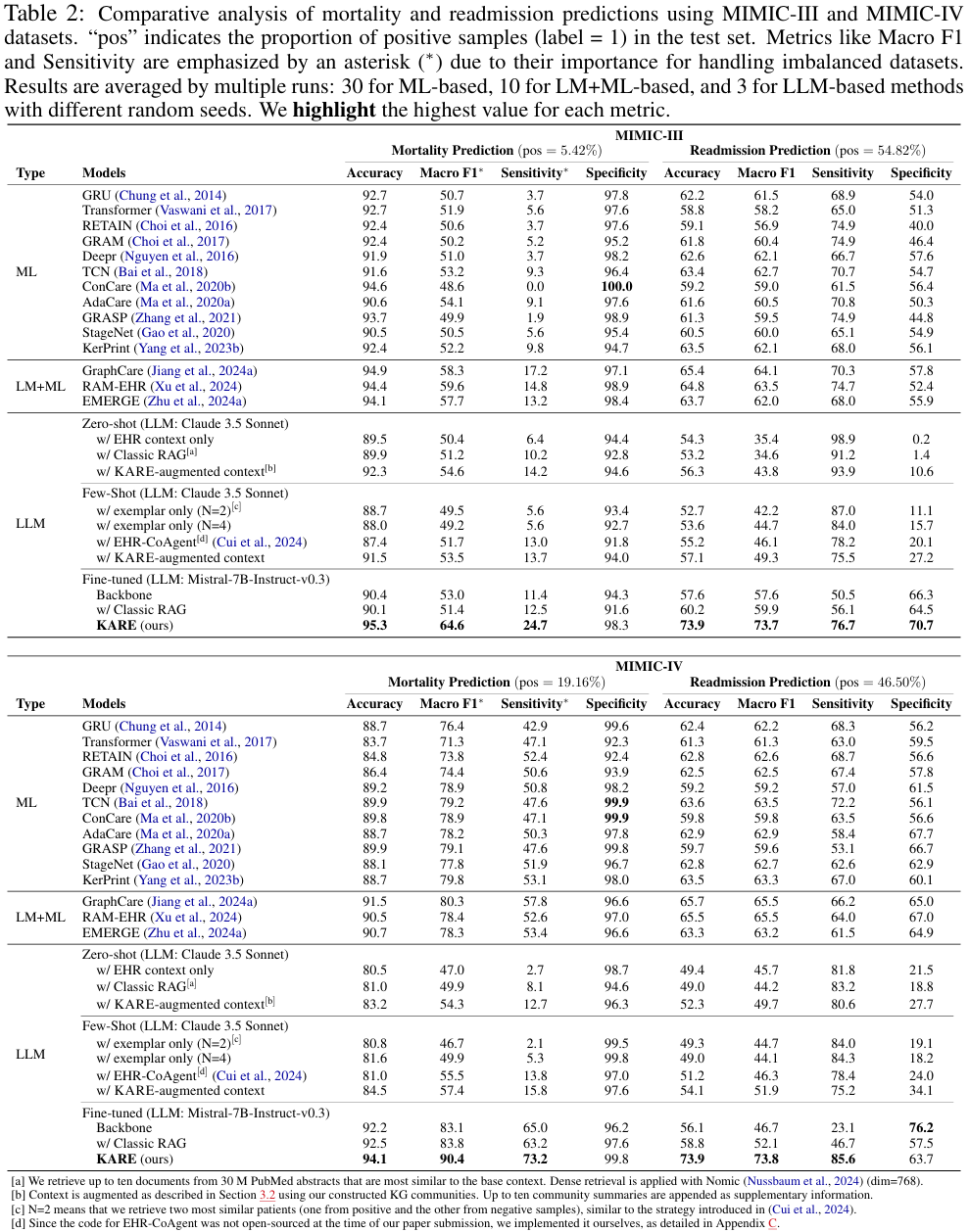
大量的实验表明,在 MIMIC-III 和 MIMIC-IV 数据集上的死亡率和再入院预测任务中,KARE 的性能比领先模型高出 10.8-15.0% 和 12.6-12.7%。 除了其令人印象深刻的预测准确性,KARE的框架还利用了 LLMs 的推理能力,增强了临床预测的可信度。
2. KARE: 知识感知的推理增强框架
KARE 框架旨在通过将相关医学知识与 LLMs 的推理能力相结合来改进医疗保健预测。 这通过以下步骤实现:(1) 医学概念知识图谱的构建和索引,(2) 患者上下文的构建和增强,以及 (3) 推理增强的精准医疗保健预测。具体如下图所示:
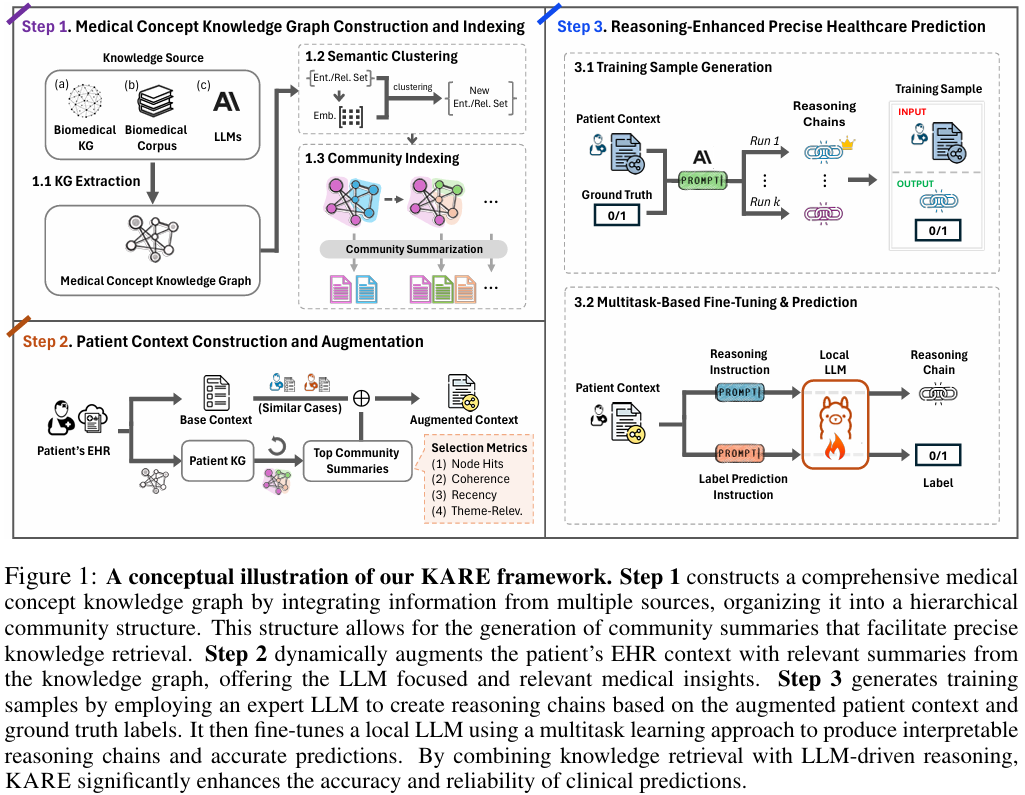
2.1 步骤1:医学概念知识图谱的构建和索引
步骤1的目标:创建一个专门为电子健康记录(EHR)数据量身定制的医学知识库。 与大多数现有的静态且未与 EHR 数据连接的医学知识图谱不同,KARE 动态生成一个高质量的知识库,可用于后续阶段的信息检索和预测。
2.1.1 医学概念特定的知识图谱提取
对于 EHR 编码系统中的每个医学概念 ,KARE从三个来源全局地为 EHR 数据集量身提取一个概念特定的知识图谱 :
(a)生物医学知识图谱 (例如, UMLS): 对于 EHR 数据中的每个医学概念 ,KARE首先遍历患者 EHR 数据集,收集每个患者数据中共同出现的前 个概念,形成相关概念集 ,从而提取一个子图 。 之后,KARE在知识图谱中为 中的每对 找到最短路径 ,并指定最大路径长度。 是通过组合所有这些最短路径构建的,其中 和 分别是所有 中节点和边的并集。
(b)生物医学语料库 (例如, PubMed): KARE遍历 EHR 数据集,对于患者的每次就诊,收集所有涉及的医学概念。 然后,根据这些医学概念从语料库中检索前 个文档。 对于每个检索到的文档,执行实体提取和关系提取以提取 KG 三元组。 提取的三元组随后被添加到文档中提到的医学概念的 中。 通过这样做,为每个概念 构建了 。
(c)大型语言模型 (LLMs): KARE遍历 EHR 数据集,并提示 LLM 识别那些有助于临床预测的概念之间的关系,其允许 LLM 在两个概念之间添加中间关系。
最终的概念特定 是来自每个来源的子图的并集:
最后,整合 EHR 编码系统中所有医学概念的概念特定知识图谱。
最终的知识图谱 定义为 ,其中 是指定 EHR 编码系统中所有医学概念的集合。
2.1.2 语义聚类
KARE采用带有自动确定的最优阈值的凝聚聚类(agglomerative clustering) 解决了知识图谱中来自不同来源的、可能指代同一概念的实体和关系命名不同的挑战。
首先,使用 LLM 为每个实体 生成文本嵌入 ,为每个关系 生成文本嵌入 。 为了确定实体和关系的最佳聚类阈值 和 ,参考了轮廓分数(silhouette score) ,该分数同时考虑了簇内相似性和簇间相异性。 KARE对实体和关系的一个子集进行抽样,使用不同的距离阈值执行凝聚聚类,并选择那些产生最高分数的阈值。
然后,KARE使用各自的最优阈值对所有实体和关系进行聚类。 每个簇由最接近簇中心的元素表示,该中心由簇内所有元素的平均嵌入确定。
最后,创建了原始实体/关系与其簇代表之间的映射 和 。 原始 KG 中的每个三元组 被映射到其对应的簇代表 ,从而得到一个精炼的知识图谱 。
2.1.3 层次化知识图谱社区检测和索引
KARE使用莱顿算法(Leiden algorithm) 将精炼后的知识图谱(KG)组织成一个从粗到细的多层次社区结构。 通过使用不同的随机性参数多次运行该算法,以探索多样的社区结构,并为每个社区生成多主题摘要,从而提供对 KG 中所含知识的更全面的理解。 为了保持计算的可管理性,限制了每个社区的最大三元组数量 和每个初始摘要的最大三元组数量 。
对于每个社区,KARE使用一个 LLM生成两种类型的摘要:
通用摘要: 对社区中的医学概念和关系进行简明扼要的概括,不侧重于任何特定主题。
主题特定摘要: 如果相关,摘要将重点说明社区中的知识如何与特定主题(例如,死亡率预测)相关。
摘要过程取决于社区的大小:
对于小社区 ,直接摘要所有三元组。
对于大社区 $(Z_{s}<size\\le z_{c})$,将三元组随机打乱并分割成子集,对每个子集进行摘要,然后迭代地聚合这些摘要,直到得到一个单一的综合摘要。<="" section="">
对于非常大的社区 ,由于 LLM 上下文窗口的限制,不生成摘要。
当从细粒度层级向粗粒度层级移动时,来自小社区的三元组被合并到更大的社区中,然后使用相同的过程进行摘要。 结果是一个不同粒度层次的社区结构,每个社区都有主题特定的摘要。
2.2 步骤2:患者上下文的构建和增强
步骤2的目标: 此步骤构建附有高度相关和细粒度医学知识的患者 EHR 上下文。
2.2.1 基础上下文构建
对于一个患者 ,KARE使用标准化模板构建其 EHR 数据的基础上下文 。 此上下文侧重于 (1) 任务描述,(2) 患者在不同就诊中的状况、程序和药物,以及 (3) 与目标患者相似的患者。 对于 (3),根据 EHR 相似性从参考集(即训练数据)中检索两个最相似的患者,其中一个与患者 具有相同的标签,另一个具有不同的标签 。
2.2.2 上下文增强
首先,通过聚合患者 EHR 中所有医学概念 的概念特定图 (在公式1中定义)来构建一个患者特定的知识图谱 ,使用了§2.1.2中的映射 和 :
从 派生出两组节点: ,代表直接出现在患者 EHR 中的医学概念,以及 ,包含 中的其余节点。
然后,为每个社区 引入一个组合相关性得分,以选择最相关的摘要进行上下文增强:
在公式3中, 和 通过比较社区 中的节点与相应的直接和间接节点集,计算直接和间接节点命中的归一化计数。 参数 用于权衡间接命中相对于直接命中的重要性。
衰减函数 将社区 中先前命中节点的贡献降低一个因子 ,其中 是一个衰减常数, 是先前选择中节点 的命中次数,仅考虑直接节点 。
其他因子定义如下:
这里, 表示文本嵌入函数, 是嵌入之间的余弦相似度, 返回节点v的就诊索引, 控制这些指标的权重。
集合 包含主题 的代表性术语(例如,对于死亡率预测,为{end-stage, life-threatening....}),与用于注意力初始化的术语相同。 公式3中提出的指标服务于不同目的:节点命中 确保了对患者状况的特异性,衰减因子促进了多样性,一致性使所选摘要与患者的整体上下文保持一致,时近性优先考虑了更新的信息,而主题相关性则维持了任务导向的选择。
所以,公式3还可以理解为:
核 心 命 中 多 样 性 惩 罚 一 致 性 时 近 性 主 题 相 关 性
2.2.3 动态图检索与增强
此外,KARE提出了一种动态图检索与增强(DGRA)方法,以迭代地选择最相关的摘要来增强患者的上下文。 在每次迭代中,它执行如下操作:

该过程持续进行,直到选择了 个摘要。最终的增强患者上下文 是通过将基础上下文 与选定的摘要连接而获得的。
2.3 步骤3:推理增强的精准医疗保健预测
步骤3的目标: 此步骤训练一个 LLM,使其能够在使用前一步构建的增强患者上下文的同时,生成推理过程并预测医疗结果。
2.3.1 训练样本生成
KARE采用一个 LLM 为每个患者 和任务 生成统一格式的推理链。此过程涉及将 (1) 任务描述 ,(2) 增强的患者上下文 和 (3) 相应的真实标签 输入到 LLM 中。LLM 生成 K 个推理链 及相应的置信度水平。KARE选择具有最高置信度的推理链,确保只使用最可靠的解释。每个患者-任务对的最终训练数据集为 ,其中 是具有最高置信度水平的推理链。
2.3.2 基于多任务的微调和预测
KARE微调一个相对较小的本地LLM(例如,一个7B参数的模型),以为每个患者 和医疗保健预测任务(如死亡率或再入院预测)执行推理链生成和标签预测。该模型使用包含任务描述 和增强的患者上下文 的输入进行训练,并在输入前添加一个指令,指示是生成推理链还是预测标签。这些输入和输出根据附录中图17所示的模板进行格式化。
在微调期间,当指令为生成推理链时(带有前缀[Reasoning]),模型会将其输出与从前一步获得的推理链 对齐。当指令为预测标签时(带有前缀[Label Prediction]),它会将其输出与真实标签 对齐。通过最小化这两个任务的交叉熵损失,鼓励开发共享表示,以增强推理和预测的性能。
在预测阶段,对于一个新患者 和任务 ,KARE向微调后的模型提供 和适当的指令。根据指令,模型可以生成推理链 或预测标签 。
3 实验
3.1 任务描述
死亡率预测。 此任务旨在预测下一次就诊的死亡结果,定义为 ,其中 是患者在就诊 期间的存活状态。
再入院预测。 此任务旨在预测患者是否会在 天内再次入院,定义为 ,其中 , 是就诊 的时间戳,并且如果 则 ,否则为0。本研究中 设置为15。
3.2 数据集
KARE利用公开可用的MIMIC-III (v1.4) 和 MIMIC-IV (v2.0) EHR数据集,并使用PyHealth进行预处理。MIMIC-III(全集)使用与GraphCare相同的方法进行处理。对于MIMIC-IV死亡率预测,保留了2152名标签为1(死亡)的患者,排除了超过10次就诊的54名患者。然后,随机(种子=42)抽样标签为0的独立患者,每位患者就诊次数不超过10次,直到样本量达到10,000。对于MIMIC-IV再入院预测,随机(种子=42)选择了5,000名标签为1(将再入院)和5,000名标签为0的独立患者。两个数据集都按患者以0.8/0.1/0.1的比例划分为训练集、验证集和测试集,确保同一患者的所有样本都仅存在于一个子集中,以防止数据泄露。使用临床分类软件(CCS)进行疾病/操作映射,使用解剖学治疗学化学分类系统第三级(ATC3)进行药物映射。
3.3 评估指标
采用四种标准的二分类指标:(1) 准确率(Accuracy),衡量整体正确预测;(2) 宏平均F1 (Macro-F1),为不平衡数据集提供平衡的度量;(3) 敏感性(Sensitivity),量化模型识别高风险患者的能力;以及(4) 特异性(Specificity),评估识别低风险患者的准确性。关于指标选择和计算的详细讨论在附录E中提供。
3.4 实验结果
[1] Müllner D. Modern hierarchical, agglomerative clustering algorithms[J]. arXiv preprint arXiv:1109.2378, 2011.
[2] Jiang P, Xiao C, Wang Z, et al. TriSum: Learning Summarization Ability from Large Language Models with Structured Rationale[C]//Proceedings of the 2024 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies (Volume 1: Long Papers). 2024: 2805-2819.
[3] Traag V A, Waltman L, Van Eck N J. From Louvain to Leiden: guaranteeing well-connected communities[J]. Scientific reports, 2019, 9(1): 1-12.
[4] Cui H, Shen Z, Zhang J, et al. Llms-based few-shot disease predictions using ehr: A novel approach combining predictive agent reasoning and critical agent instruction[C]//AMIA Annual Symposium Proceedings. 2025, 2024: 319.
安全学术圈招募队友-ing
有兴趣加入学术圈的请联系 secdr#qq.com
声明:本文来自安全学术圈,版权归作者所有。文章内容仅代表作者独立观点,不代表安全内参立场,转载目的在于传递更多信息。如有侵权,请联系 anquanneican@163.com。
